6月28日,中国科学院北京生命科学研究院计算基因组实验室赵方庆教授团队在国际知名杂志Nature Communication发表论文,利用环状RNA预测分析结合长片段测序技术,首次实现了针对环状RNA可变剪切状态进行了系统研究(Gao et al., 2016)。本文的技术思路比较新颖,对于环状RNA功能研究有很高的借鉴价值。下面我们就一起剖析一下这篇力作的精妙之处吧。
本文讲述了什么样的故事?
RNA结构预测是RNA研究非常重要的工具,在mRNA以及lncRNA等RNA研究方向中均有非常好的运用。与此同时,线性RNA分子中经常存在的可变剪切(Alternative splicing, AS)也非常常见。但这类RNA研究的思路和角度却很少在环状RNA的研究中报道。环状RNA本身就是可变剪切的产物,但每个环状RNA分子内部是否仍有可变剪切方式?环状RNA的二级结构如何?这些都是非常有趣的问题。
赵方庆教授团队基于该问题,设计了一种新的整合环状RNA结构与序列信息的技术分析流程,作者称之为CIRI-AS(下面会有详细的介绍)。作者在10种人类细胞以及62个果蝇不同组织来源的样品中分析了环状RNA中存在的可变剪切。最终证明存在四大类环状RNA普遍存在的可变剪切方式,大大增进了对环状RNA的认识,也为环状RNA研究提供了新思路和新工具。
CIRI-AS技术流程是怎么进行的?
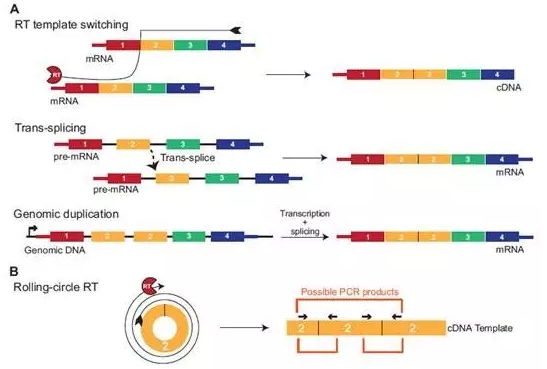
传统的基于RNA-Seq的环状RNA组学分析策略瞄准的是与基因组序列比对分析出来的反向拼接位点(环化位点),绝大部分环状RNA鉴定相关的算法是基于这一认识设计的。本文作者提出了新的思路:环状RNA分子除了“环化位点”,有没有别的序列上的特征?包括有没有像所对应的线性RNA中经常存在的可变剪切形式?这个问题之前还没有引起学术界的关注。作者基于该问题,创造性的提出了在原有的分析“环化位点”(BSJ)的基础上,分析所涉及的Reads中是否存在可变剪切的位点(forward-spliced junctions,FSJ)。
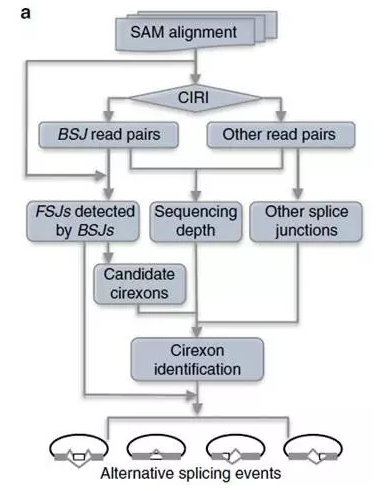
图1 CIRI-AS技术流程
四种可变剪切方式在环状RNA中都能找到,但存在比例差别较大:
理论上存在可变剪切的外显子有四种可能的可变剪切方式:跨越外显子(exon skipping, ES)可变的5’或3’拼接位点(alternative 5’or 3’ splicing site , A5SS and A3SS) 以及保留内含子(intron retention, IR)。作者通过分析10种人类细胞以及62种果蝇的不同组织样品中环状RNA可变剪切的特征,汇总得出结论:四种理论上可能的可变剪切方式在环状RNA中均存在,如下图所示。有趣的是,文中的结果表明四种形式的可变剪切的存在比例还是有挺大差别的,最常见的是ES,而IR形式的存在比例最低。文中还分析了四种类型可变剪切的比例情况,发现在环状RNA中存在的可变剪切与所对应的mRNA中的情况有较大差别,说明环状RNA的可变剪切调控方式与mRNA有所不同。
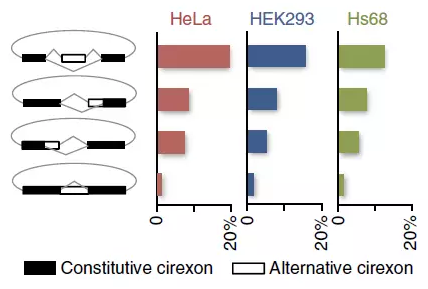
图2 环状RNA中存在四种可变剪切方式,但比例相差很大
可变剪切给环状RNA检查带来的困扰:
相信很多环状RNA研究的人都遇到过用PCR进行环状RNA反转扩增的时候经常回出现多条条带的问题。之前Julia Salzman等曾发表综述文章,分析了各种理论上可能的导致环状RNA扩增引物出现多条条带的问题,他们分析认为有可能是出现了RNA模板切换,线性RNA的可变剪切甚至是基因组水平的局部序列多倍化突变导致的,也有可能是环状RNA本身因为没有被降解而出现滚环性反转而导致cDNA序列延长。
本文的结果为该现象提出了另一种新的解释:环状RNA本身就存在可变剪切,因此导致PCR扩增产物出现多条带。作者就从反转录PCR实验中直接证明了这个可能性,而且得到测序结果的直接证据。因此大家在研究环状RNA的过程中还是建议尽量扩增并克隆出电泳产生的条带进行测序鉴定,以确认是那种可能性导致的多条带问题。
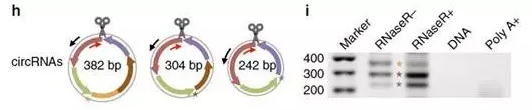
图4 环状RNA中存在的可变剪切方式也会导致PCR多条带现象
环状RNA中可变剪切外显子RBP结合位点与mRNA有较大差别:
从可变剪切存在比例的结果来看,环状RNA中可变剪切的方式与mRNA有较大差别,作者进一步分析了这些可变剪切对应的外显子中RBP结合位点的特征,发现环状RNA内可变剪切外显子中的位点信息与mRNA差别非常大,例如QKI, Tra2b以及hnRNPU的结合位点在环状RNA的可变剪切外显子中非常高,而mRNA中相对较低。

图5 RBP结合位点在环状RNA和mRNA的可变剪切外显子中分布差别较大
环状RNA可变剪切方式存在更多样化的细胞定位及细胞类型特征:
作者比较了三种人类肿瘤细胞(HeLa-S3, HepG2, K562)和四种非肿瘤细胞(H1-hESC, GM12878, HUVEC 和 NHEK)中环状RNA可变剪切的细胞定位分布情况,发现四种可变剪切类型的环状RNA均更倾向于定位于细胞核中,在不同的细胞系中存在高度的多样性特征。(PS:山人以为,这一发现表明环状RNA的可变剪切形式很可能隐含着重要的生物学功能,非常值得深入研究)。
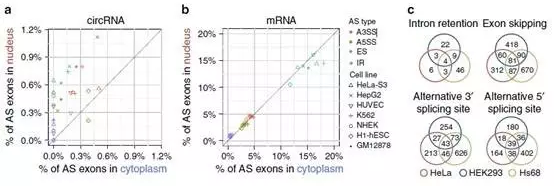
图6 环状RNA可变剪切的细胞定位和细胞系特异性特征分析
环状RNA可变剪切呈现组织和发育状态特异性分布特征:
环状RNA可变剪切反式在细胞定位和培养细胞类型方面的特征非常明显,那么很自然的,环状RNA的可变剪切是否存在组织和发育特异性?作者分析了果蝇中不同组织及发育阶段的样品中环状RNA可变剪切的情况,结果表明存在三种情况:有一部分环状RNA的可变剪切是普遍存在于所有细胞和组织来源的样品的,有趣的是有一类是特异性存在于中枢神经系统的,剩下的则没有明显的组织和细胞特异性特征。
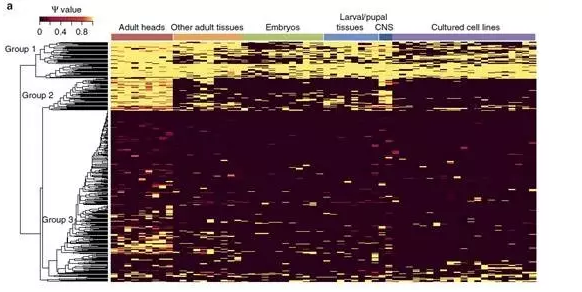
图7 果蝇组织特异性环状RNA可变剪切分析
这一类中枢神经系统特异性的可变剪切引起了作者的兴趣,分析了这些基因所对应的通路:
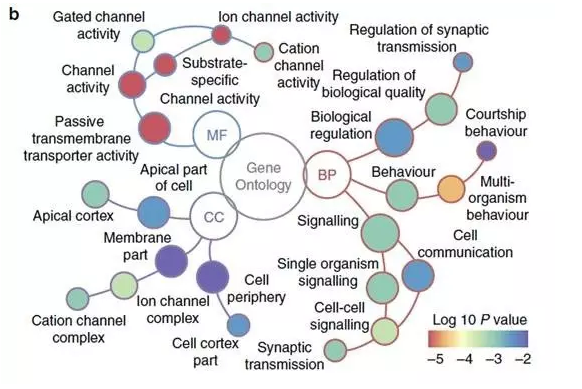
图8 中枢神经系统特异性环状RNA可变剪切对应基因的通路分析(BP: biological process; CC: cellular component; MF: molecular function)
总结:
本文为我们展示了一个全新的环状RNA研究思路,是一项非常有创新价值的研究成果。 此前,环状RNA中携带内含子的情况早有报道,文献中称此类环状RNA为EIciRNAs,这类环状RNA通常定位于细胞核内,会影响对应基因的转录过程。从本文的结果来看,不仅仅是EIciRNAs(对应于本文的可变剪切类型为IR),其余形式的环状RNA似乎也更倾向于定位于细胞核内,至于发挥什么功能还是未知的谜题。拜读本文的过程中,山人收到了很大的启发,在此跟诸位一起分享一下:
1. 长度长RNA-Seq在环状RNA的研究中非常必要
CIRI-AS技术流程的优势是不仅仅关注传统环状RNA分析位点,还分析了相关Reads中可变剪切的信息。该技术流程要发挥更大的作用,需要基于长读长的RNA测序平台,也需要更高的测序深度。从文中的数据来看,要实现对样本的有效检测,测序通量少则12Gb,最高甚至需要40Gb的通量。但相信随着高通量测序技术的发展,这些技术要求都不是问题。

图9 环状RNA可变剪切分析实验测序通量
2. 环状RNA分子中可变剪切的生物学意义是环状RNA功能研究的新切入点
从本文的结果来看,环状RNA的可变剪切存在细胞定位,组织类型等多方面的特异性特征,尤其是作者最后筛选到的在果蝇中枢神经系统中特异性存在的一些环状RNA可变剪切对应的基因非常有研究价值。
本文以及之前的诸多重量级的环状RNA研究论文中很大比例的都是从组学的角度进行研究的。组学有利于全面系统的认识一类分子的表达和定位等特征,往往“只见森林,不见树木”,筛选得到一批特征明显的环状RNA分子,但这些分子如何发挥功能的问题仅靠组学的思路无法回答,必须瞄准特定基因进行深度挖掘分析。
组学水平探索可变剪切形式需面临测序读长有限等问题,目前技术条件还有不少的局限性。与此不同,如果研究特定基因的环状RNA及对应的线性RNA中可变剪切形式,分布和定量则相对容易得多。只需要通过合理设计筛选鉴定出这些RNA分子的序列,就可以更清晰的了解它们的序列,分布和定量等问题,结合感兴趣的生理或病理过程,是非常好的研究RNA表达特征的生理病理功能的思路。
当然,以上仅仅是山人在拜读这篇文章的时候想到的而已,没有经过严谨的论证和同行间讨论,仅为个人见解。
参考文献:
Barrett, S.P., and Salzman, J. (2016). Circular RNAs: analysis, expression and potential functions. Development 143, 1838-1847.
Gao, Y., Wang, J., Zheng, Y., Zhang, J., Chen, S., and Zhao, F. (2016). Comprehensive identification of internal structure and alternative splicing events in circular RNAs. Nat Commun 7, 12060.





来第一个抢占沙发评论吧!