环状RNA(circular RNA,circRNA)不同于传统意义上的线性RNA分子,是一大类具有闭合环状结构的特殊非编码RNA。circRNA由于其闭合环状结构,不易受RNA外切酶影响,因此不易降解,表达更稳定,这使得环状RNA在作为新型临床诊断标记物的开发应用上具有明显优势。对于circRNA的高通量筛选,实践证明,基因芯片是一个非常好的选择。
当然,基因芯片只是前期的筛选平台,当完成这一步后,怎么进行qRT-PCR验证呢?circRNA的定量验证到底和线性RNA(mRNA/lncRNA)有什么不同的地方呢?让我们一起来看看。
circRNA验证引物设计原则
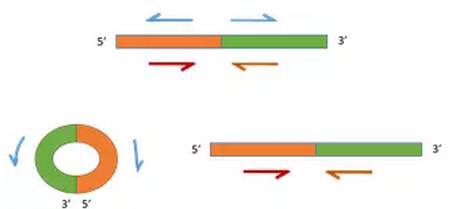
circRNAs区别于线性RNA的原因在于circRNA来源于RNA转录本的反向剪接(backsplice)。因此,circular RNA验证时采用和线性mRNA分子引物方向相反的方式(Divergent PCR),用扩增区域覆盖剪接位点的反向引物能扩增出来证明其特异性表达。
备注:蓝色引物用于特异性检测circRNA,红色引物用于特异性检测线性RNA。
circRNA引物设计序列的获取
以SBC human circRNA array为例,其探针长度为60 nt,包含circRNA剪接位点。因此,从反向引物设计原则和引物与探针匹配的原则进行引物设计。具体分析过程如下:
做完芯片后,公司提供差异circRNA的全序列信息,如:
hsa_circ_0000027在芯片注释表中的探针序列(芯片结果表中直接给出):
通过将探针序列与对应circRNA全序列的比对,可以看出探针序列为circRNA全序列的末位序列与起始序列反向拼接而成。拼接点的两个探针GG分别为hsa_circ_0000027的头和尾碱基(剪接位点)。设计验证引物时,先构建剪接位点左右150 bp的线性序列,如下:
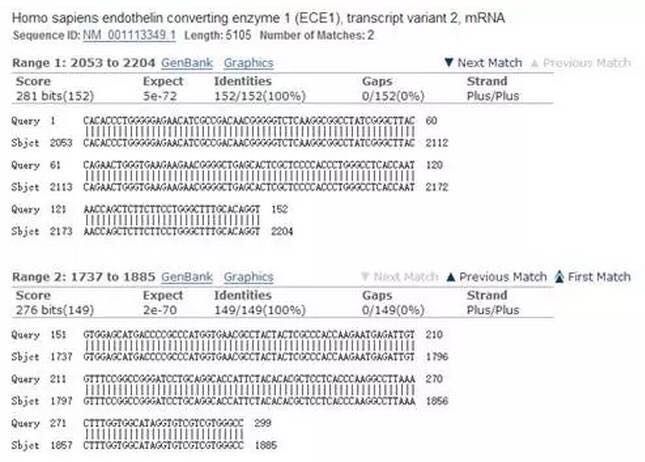
以上300 nt的序列就是以剪接位点为界限,上下游各取150 nt的序列反向拼接而成。为验证该序列的特异性,可通过NCBI进行BLAST,确认其唯一性。
从NCBI结果可以看到,该序列是ECE1的两个不连续完整片段,因此,是来源于ECE1特有的circRNA。如果提供的circRNA全长序列小于300 nt,可通过首尾拼接构建包含剪接位点的线性序列用于定量引物设计。
反向拼接获得的引物设计序列可以直接按照常规的mRNA设计原则进行引物设计。
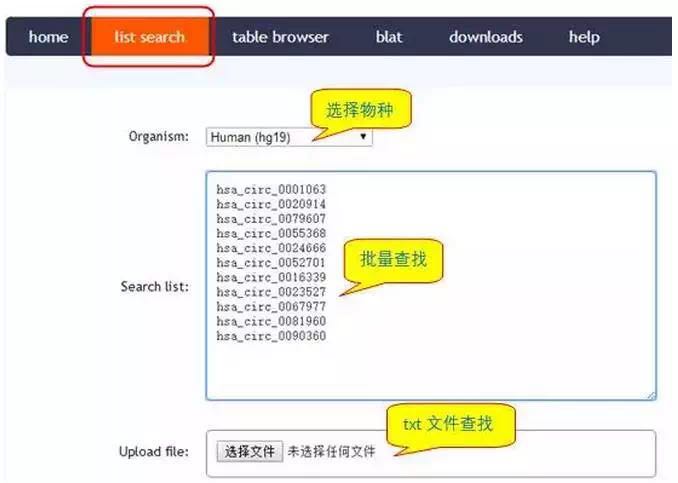
此外,您也可以通过数据库circBase(http://circbase.org/)获取circRNA的序列信息,具体方法如下:
点击search即可下载circRNA序列。
批量circRNA序列查找:
点击search,按上述方法即可批量获得circRNA序列。
总结
circRNA定量验证的关键在于反向引物的设计,其核心是构建包含反向剪切位点的引物设计序列。获得引物设计序列后,即可按mRNA的引物设计原则进行引物设计,上下游引物因设计在剪切位点的两侧,确保扩增序列包含剪切位点。CircRNA的qRT-PCR引物设计,您是否已经GET到了?








你好,我想问一下为什么是150bp?200bp可以不?
你好,通过Circbase数据库export出来的fasta格式spliced后的具体序列是已经确定的?还是只是预测序列,还需要实验加以测序验证才能确定?
测序得到的序列,也是需要验证的,但是绝大部分都是对的
您好,我想问一下,我想设计跨接口的下游引物一般跨越接口几个碱基比较合适,还是无所谓?我们是用芯片筛选出的circRNA,那是否可以直接选用探针序列作为跨接口的引物序列呢?
请问你用探针序列作为跨接口的引物序列了吗?