原创作者: 天地本宽 | 生信技能树
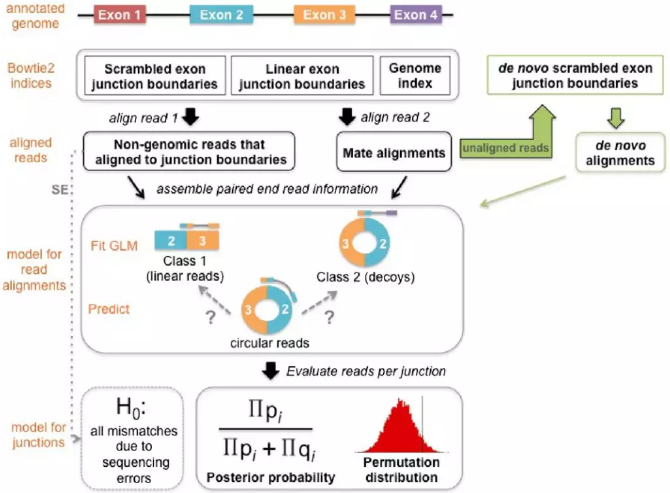
根据circRNA分析软件benchmark文献推荐,KNIFE准确度最高,circRNA分析优先推荐使用KNIFE工具。
参考文献:A comprehensive overview and evaluation of circular RNA detection tools
1. 下载修新版KNIFE软件
从release中选择v1.4稳定版本下载,不建议使用git clone下载,开发版还有一些bug未解决。
cd ~/biosoft/
wget https://github.com/lindaszabo/KNIFE/archive/v1.4.tar.gz
tar zxvf v1.4.tar.gz && cd KNIFE-v1.4
cd circularRNApipeline_Standalone/analysis
# 添加执行权限,否则运行过程中会由于部分软件无执行权限报错。
sudo chmod a+x *
2. 确保所有R、python、和shell脚本可以执行
如果无法运行./completeRun.sh,需要将sh默认执行程序设置为非dash
sudo dpkg-reconfigure dash
3. 安装下面软件,并添加至环境路径
Bowtie2 2.2.1+ (tested with 2.2.1)
Bowtie 0.12.7+ (tested with 0.12.7)
perl
python 2.7.3+ (tested with 2.7.5) and the numpy and scipy libraries
R 3.0.2+ (tested with 3.0.2) and the data.table package
samtools 1.2+ if you want alignment files output as bam instead of sam files
4. 下载bowtie索引文件
a. 创建索引文件夹
创建以下两个文件夹,分别用于存放bowtie和bowtie2索引文件。
circularRNA/pipeline_index和 circularRNA/pipeline_denovo_scripts/index
b. 下载bowtie索引文件并解压到相应文件夹
Human (hg19), Mouse (mm10), Rat (rn5) and Drosophila (dm3)有可用的junction索引文件下载。
作者同时打包了所有的转录组、基因组和核糖体RNA索引、fasta文件和gtf文件。
需要将bowtie1索引文件解压至以下文件夹:
cd /path/to/circularRNApipeline_*/denovo_scripts/index
tar zxvf genomeId_BT1index.tar.gz
将bowtie2索引文件解压至以下文件夹:
cd /path/to/circularRNApipeline_*/index
tar zxvf genomeId_BT2index.tar.gz
gtf文件需要下载并解压缩至以下文件夹:
cd /path/to/circularRNApipeline_*/denovo_scripts
gunzip genomeId_genes.gtf.gz
KNIFE提供的hg19参考基因组和索引文件可以通过下面链接下载: https://mega.nz/#F!RtsCHCQb!fyxYNWjoCef5Ie361vUxiA
如果内存够大,也可以用自己的机器构建索引。
5. 按照testOutput/README指导进行测试
(1)下载release1.1中提供的两个fastq.gz文件
cd /path/to/KNIFE/testData
nohup wget -c https://github.com/lindaszabo/KNIFE/releases/download/v1.1/SRR1027187_1.fq.gz &
nohup wget -c https://github.com/lindaszabo/KNIFE/releases/download/v1.1/SRR1027187_2.fq.gz &
(2) 测试KNIFE是否安装好(单机运算)
sh completeRun.sh /home/jshi/biosoft/KNIFE/testData/ complete /home/jshi/biosoft/KNIFE/new testData 8 phred64 circReads 40 1 2>&1可以将输出文件和github中给出的结果进行比较,看是否完成了环状RNA分析。
值得注意的是,全部分析流程都被包装到了一个shell脚本里。





来第一个抢占沙发评论吧!