原创作者: 土豆力| 生信草堂
本篇为植物circRNA系列文献阅读第三篇,接下来几篇内容都将围绕模式植物拟南芥circRNA数据型文章报道,通读以后能够快速了解不同的circRNA数据常规分析流程。今天分享的文章题目为:Genome-Wide Identification of Circular RNAs in Arabidopsis thaliana,文章链接为 https://doi.org/10.3389/fpls.2017.01678。
CircRNA的概括
1. 非编码RNA涵括多种功能RNAs,例如转运RNA(tRNA)、核糖体RNA(rRNA)、microRNA(miRNA),小干扰RNA(siRNA)、反式作用小干扰RNA(tasiRNA),小核RNA(snRNA)、小核仁RNA(snoRNA)和非编码RNA(ncRNA),其中非编码RNA又可以分为长链非编码RNA (lncRNA)及环状RNA(circRNAs)。
2. 环状RNA (circRNA)是一类由mRNA前体经反向可变剪切而来,3’和5’端共价闭合的单链转录本,根据组成来源,circRNA分为外显子circRNA、基因间circRNA和内含子 circRNA。
3. circRNA序列及表达通常保守,且具有细胞类型特异性表达以及组织特异性,通过竞争性结合miRNA或者蛋白参与转录后水平调控。在特定条件下,circRNA具有编码能力,部分circRNA产物能够参与调节可变剪切过程。
主要材料与方法
1、数据来源
根据前人研究结果,拟南芥生长周期能够分为以下七个阶段:
阶段1.04(4个莲座叶长度> 1毫米);
阶段1.14(14个莲座叶> 1毫米长);
阶段3.90(莲座生长完整);
阶段5.10(出现第一个花蕾);
阶段6.50(50%的花蕾已绽放);
阶段8.00(第一个果荚开裂);
阶段9.70(完全衰败;种子成熟可收获)。
14个拟南芥链特异性文库分别来自于(详细样本编号见原文表1):
a. 七个阶段的整株植物混样;
b. 4个阶段(阶段1.14、阶段3.90、阶段6.50、阶段8.00)的三整株植物;
c. 阶段6.50中,5个组织(根、茎、叶、花、果荚)样本;
d. 阶段3.90时,3个处理(300 mM甘露醇(干旱),200 mM NaCl(盐度)和38°C(热))1个对照,处理12小时后地面上植物样本,并设对照组。
构建,其中混样样本1采用PE125策略, Illumina HiSeq 2500平台测序,其他13个样本采用PE150策略, Illumina HiSeq X Ten平台测序,平均测序深度达106X。NCBI SRA数据编号SRP069764。其他植物circRNA数据来源PlantcircBase。
2、分析流程
1. 使用NGS QC Toolkit v2.3.3进行过滤及质控,包括接头序列、N碱基含量大于5%的读段及低质量读段(reads with base quality ≤ 20)。
2. 序列比对使用TopHat2 with BOWTIE2 (v2.0.5),参考基因组选用Arabidopsis_thaliana TAIR10.30 ENSEMBL。
3. CircRNA预测使用find_circ,参数默认,circRNA丰度预测基于接头读段计数。
4. Bedtools用来注释包含重叠区域的circRNA。
5. BLAST用于拟南芥和各植物circRNA之间的同源性搜索。
6. BLAST2GO用于circRNA来源基因的功能注释。
7. TargetFinder用于预测circRNA互作的miRNA,调控网络图使用Cytoscape 3.5.1绘制。
3、实验验证
1. PCR扩增;
2. qRT-PCR定量。
结果
1、CircRNA的鉴定及描述
14个样本总共获得187.97Gb数据,其中151.37Gb来自于13个样本,有参组装总共获得795.62 million raw reads,过滤掉接头及低质量序列最终获得763.81 million clean reads (96.00%)。通过find_circ预测,在14个样本中总共鉴定出5861条circRNA,其中包含1,275 (21.75 %) 新鉴定circRNAs。
a. 染色体定位
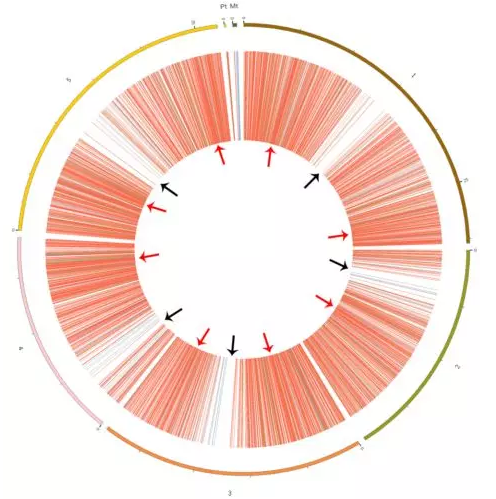
图1所示所有鉴定的circRNA染色体定位情况,包括线粒体及叶绿体染色体,不难发现,circRNA在染色体上并不随机分布,1号和5号染色体circRNA丰度较高,2、3、4号和叶绿体染色体上分布较少,而线粒体DNA上只包含1.6%的circRNA。
b. 来源组成情况
4,990 (85.1%)的circRNA由外显子构成,650(11.1%) 来源于基因间,只有221产生于内含子序列。2546条来源基因能够产生一条以上的circRNA,而其他770条来源基因只能能产生一条circRNA。
c. 保守性分析
BLAST比对分析发现,拟南芥与其他植物circRNA序列有着不同程度相似性,其中有六条拟南芥circRNA序列在水稻、番茄及玉米也出现,说明不同植物物种间circRNA序列具有保守性。
2、CircRNA的功能注释
circRNA来源基因的功能注释用于评估circRNA的潜在功能,包含GO注释、KEGG注释。miRNA靶位点预测用于评估circRNA能否通过miRNA参与转录后调控。作者发现39(0.67%)circRNA包含潜在的miRNA结合位点,其中9条包含不止一个miRNA结合位点;绝大多数的circRNA与配对的miRNA完全互补;同时不同的circRNA可能具有相同miRNA的结合位点。
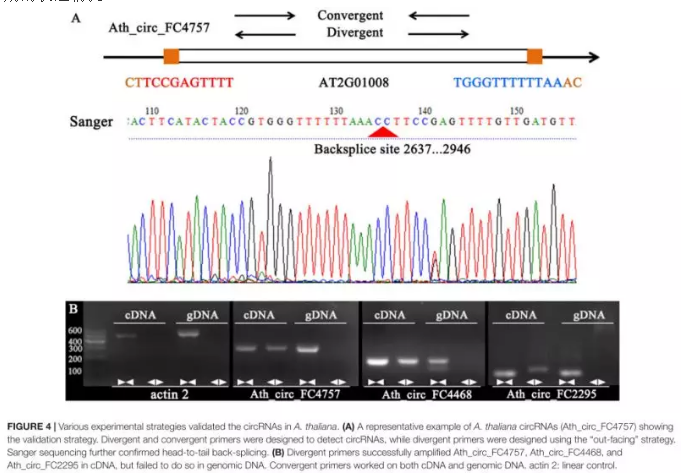
3、CircRNA的验证
PCR验证的思路主要通过设计发散性引物,通过比较cDNA模板及DNA模板的扩增电泳条带及测序来说明circRNA的存在。qRT-PCR实验挑选了10条来简单说明circRNA在不同组织样本及时期的表达情况。
小结
本文为常规circRNA转录组文章。通读全文有以下几点思考:
1. 拟南芥作为一个模式植物,基因组注释信息较为完善。作者采集了多个样本进行深度测序,只是从宽度的角度初步鉴定了拟南芥环状RNA信息,其中近80%的鉴定已有报道。
2. 多个样本数据仅作为一个合集进行分析,只是做了GO和KEGG的简单可视化,得到的miRNA互作位点并没有体现出针对性,且39(0.67%)的预测miRNA结合位点量偏少。
3. 作者选取5个随机序列进行扩增,鉴定了3个环状RNA的存在。qPCR实验验证了不同生长过程中circRNA的表达量变化,但是所选对象之间并没有直接联系,后续验证了两条与光合作用相关的circRNA亲本基因的表达量变化,仅仅得到了两条序列在叶中表达而在根部几乎不表达的结论。
综上几点,文章有多处可以继续深挖的信息点,但是介于样本收集的方式且测序并没有生物学重复,因此后续分析需要更多的支持。





来第一个抢占沙发评论吧!