文章:生信草堂(微信公众号)
01 前 言
今天和大家分享一篇经典的综述,题目是 Detecting circular RNAs: bioinformatic and experimental challenges ,为circRNA研究提供一些参考。circRNA是一类新兴的非编码RNA,具有特殊的拓扑结构和稳点保守 性,而成为研究热点。
文章发于Nature Reviews Genetics (if ~ 40) 是早期比较经典的综述文章 http://dx.doi.org/10.1038/nrg.2016.114。作 者综述了实验和生信分析中识别circRNA可能出现的偏差,举例并加以讨论。同时提出circRNA在识别上还存在一些问题:
1. 虽然已有多种算法支持circRNA的识别,但是缺少对假阳性和假阴性率的评估。
2. 对于剪切位点的选择以及RNA过程的模型还不够。
02背 景
环状RNA的backsplices 大多数发生于注释的外显子边界上或者包含经典的剪切信号的位置 (spliceosome)。
大多数的环状亚型 (isoforms) 只能产生1-2个可区分的circRNA,但是也有个例。
大部分细胞中的circRNA丰度在2-4%左右,当时有些细胞类型中也会有较高水平。
已在从人类到小鼠、果蝇、蠕虫、简单的生物如真菌、植物中均检测到了circRNA,对比亿万年的进化,circRNA表达不仅保守而且经历多次独立进化。
虽然circRNA mini-gene包含核糖体嵌入位点 (IRES)启动翻译,但是非编码是circRNA普遍的规律。
03 识别剪切过程中的挑战
精确的剪切位置比对识别。
使用注释可以提高识别的精确度。
注意一些circRNA包含A-rich序列,因此对poly(A)+ RNA文库测序结果需要通过算法过滤低表达的mRNA 转录本。
04 识别circRNA中的挑战
· 实验
1. circRNA没有poly(A)尾巴,可以通过此特征进行纯化。
2. 由于RNA测序片段大小的选择,只有在接头扩增前,RNA没有被打断的情况下,可能会影响circRNA的识别。
3. 反转录模板可能会导致technical artefacts,产生假阳性。
4. 长同源序列会促进模板转换 (template switching), 对于基因产生多个共享同构外显子 (constitutive exons) 的亚型来说是一个很严重的问题。
· 生信分析
1. 单向测序可能导致反向剪切位置的来源的误判。
2. 外显子附近的简并序列产生同源性和测序错误可能导致假阳性。
3. 对于线性剪切的探测可以增加识别的敏感对,但是实际上导致了高假阳性率。
05 环状RNA识别算法的比较
双端测序、更高的读取范围可提高识别敏感度,更多样本重复、RNase消化线性RNA以及统计方法将降低假阳性。
· 不同的算法过滤机制及高可信度子集的选择标准会导致不同的结果。
· 一些无参识别circRNA的算法为了降低假阳性,只选取唯一映射的读段并检测经典的剪切位点,来排除已知的circRNA isofroms。例如find_circ,三方评估结果发现,其具有较低的敏感性并且可能会有很多假阳性报告。
· 使用相同的模拟数据,所有的算法显示可以通过增加读数 (read count)来提高敏感度但是同时会降低其识别的特异性 (specificity)。
· 套索结构 (lariat)与circRNA相似,具稳定性以及不受RNase R影响,因此也作用为circRNAs的识别标记。所有算法中,circRNA的识别少于0.17%可能与套索相联系。
Table 1 Filtering criteria for selection of high-confidence circRNAs
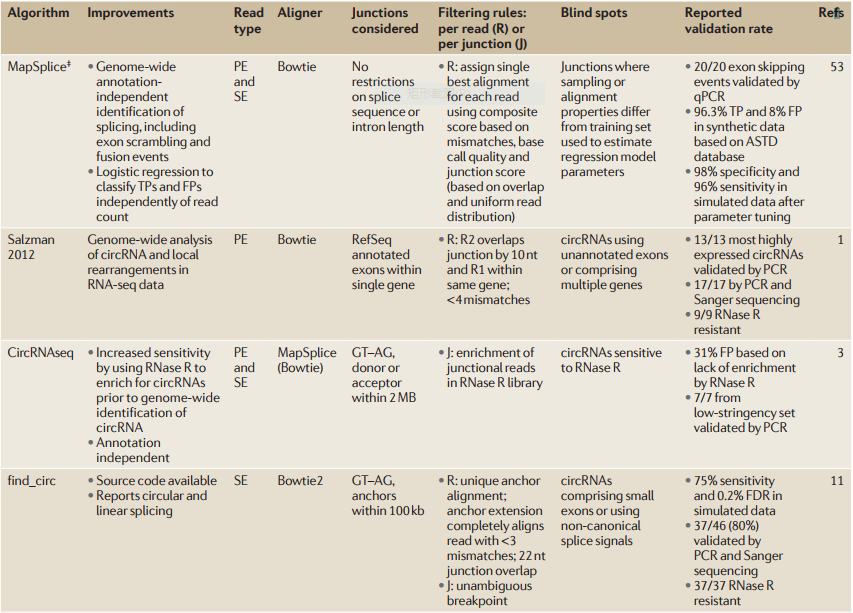
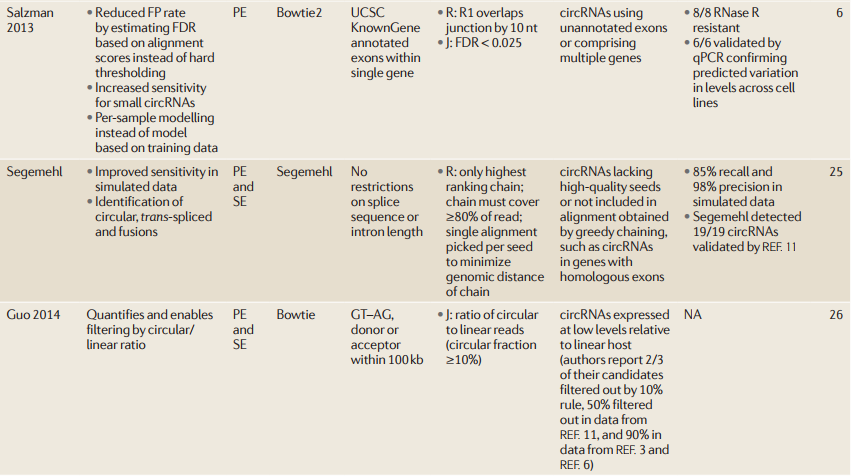
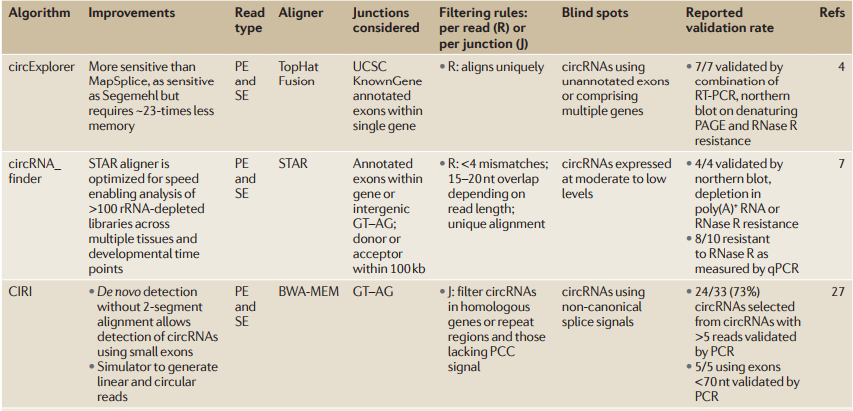
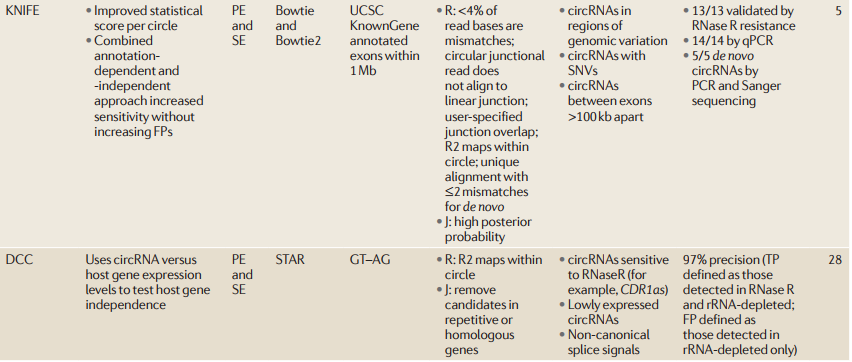
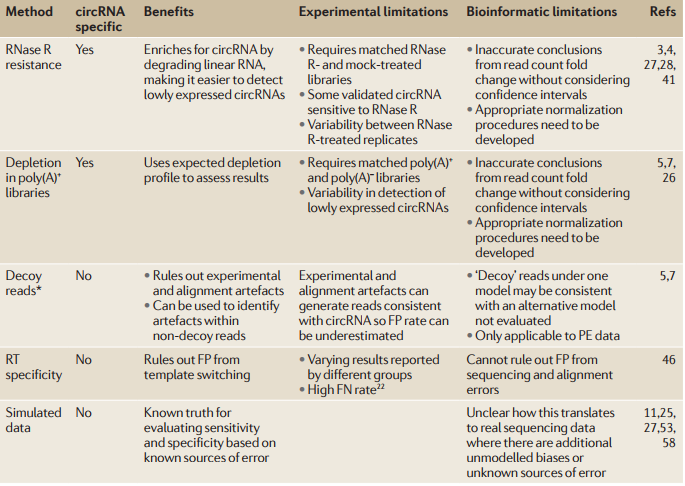
06 验证circRNA识别的讨论
· RNase R treatment
处理后的样本在识别circRNA时,可以确认假阳性的识别结果。但是在处理的过程中,可能会导致部分在文库准备过程中断裂的基因被消化,而这些被消化的基因无法判断是否与circRNA的形成相关。
对比两个文库的数据时,归一化处理比较更具有意义。
· depletion in poly(A)+ libraries
circRNA不具有poly(A)尾,但是在 poly(A)+ 文库中可能也存在着一些circRNA,它们通常表达水平低下。因此,当只有单个预测的circRNA在 poly(A)+ 文库出现,并不足以证明其正假阳性。
· decoy reads
对于circRNA来说,decoy reads 包括map到反向可变剪切上的和map到被定义为反向剪切的基因区域中的两种。由于实验以及比对方法上可能产生的人工片段干扰circRNA识别,例如外显子同源性等,decoy reads 应该选用合适的模型进行预测,并且提供统计学分值来控制假阳性。
· RT specificity
尽管缺乏RT特异性可以提供circRNA真阳性的证据,但是该实验方法无法从人工产物中区分出circRNA,可能导致高的假阳性,需要进一步实验验证。
· simulated data
模拟数据对于算法的系统局限性具有较好的评估,但是较于实验数据来说,由于生物化学事件并不完全知晓,因此模拟数据的复杂程度不及RNA测序数据。
Table 2 Methods used to assess the genome-wide accuracy of algorithms
07 建议统计检验
对于真实数据的全基因组假阳性circRNA的鉴定,对比RNase消化后的残余量指标相比,poly (A)文库中circRNA的竭尽 (depletion) 指标更合适。
对多个重复的数据进行表达分析时,每一个重复必须分别分析标准误差。支持双端测序数据进行circRNA表达的量化。
08 旁证:功能性分析
来自许多基因的高表达circRNA也具有保守性。
独立于线性转录本表达水平,circRNA具有活跃的调控模式。
09 小 结
作者从测序准备文库以及算法两个方面对circRNA的鉴定进行讨论。在文库准备上应该将circRNA尽可能的富集,例如去线性去核糖体RNA;在识别上对接头序列的真阳性和假阳性进行分类。有参识别可以进一步的提高识别精度,但是无参也会提供一系列新鉴定RNA的信息,例如非经典剪切信号等。RT模板在扩增上可能出现”人造”干扰,在算法上应该加以避免。同时强调统计方法的合理使用可以提高识别的真阳性。同时总结了circRNA识别算法的现状以及不同实验处理下对circRNA识别的影响,为深入circRNA研究提供了参考依据及建议。





哇 看到这篇文章真是太及时了 谢谢