最近的研究已经证明环状RNA(circRNA)无处不在,并且具有不同的生物发生功能和机制。在这些研究中,circRNAs的计算分析已被普遍用作提供高通量方法来检测和分析circRNA的不可或缺的方法。然而,如果没有对基本策略的全面理解,这些计算方法可能不会被适当选择或用于特定的研究目的,并且一些误解可能导致分析中的偏差。在最近发表的一篇综述中,作者说明circRNA识别的关键步骤并总结不同策略的权衡,涵盖所有流行的circRNA检测算法和各种下游分析。我们也澄清了一些常见的误解,并强调这些计算方法的应用领域。
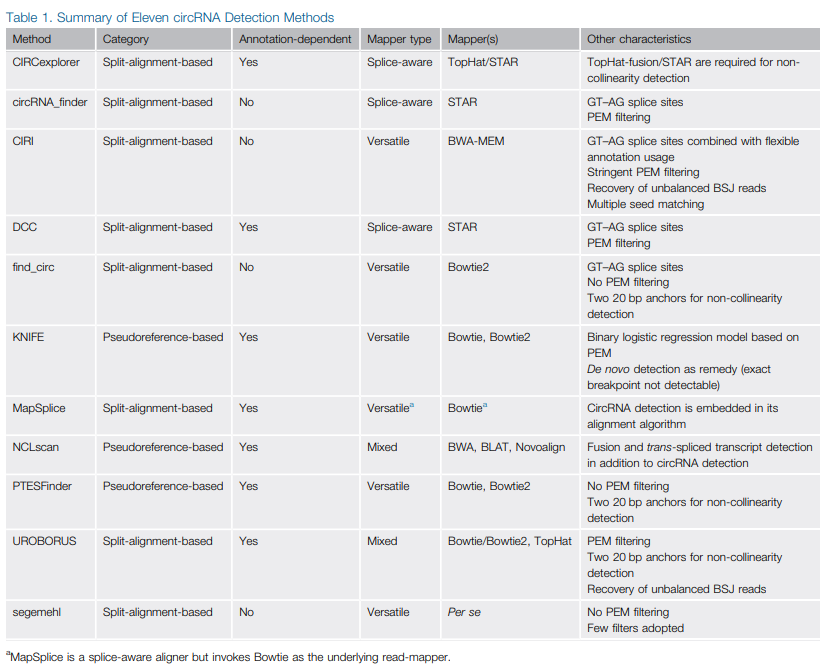
作者提出参考基因组对于所有检测算法都是必需的,但可以在检测工作流程中以不同方式使用。最常见的用途涉及所有测序read与参考基因组的直接比对, 大多数检测算法(如find_circ,CIRCexplorer ,CIRI和UROBORUS )可以归入此类别. 从比对中捕获的明显特征是称为背拼接结(BSJ)的环结, 这一类别的检测算法可以称为基于分割对齐的方法(split-alignment-basedapproaches),对于其他算法,如KNIFE和NCLscan ,将参考基因组与相应的基因组注释组合在假定的BSJ周围构建伪序列(图1C)。随后的步骤然后集中于测序read与这些伪序列的完全比对以识别BSJ读数。这个类别中的检测算法可以被称为基于伪基准的方法(pseudoreference-basedapproaches).
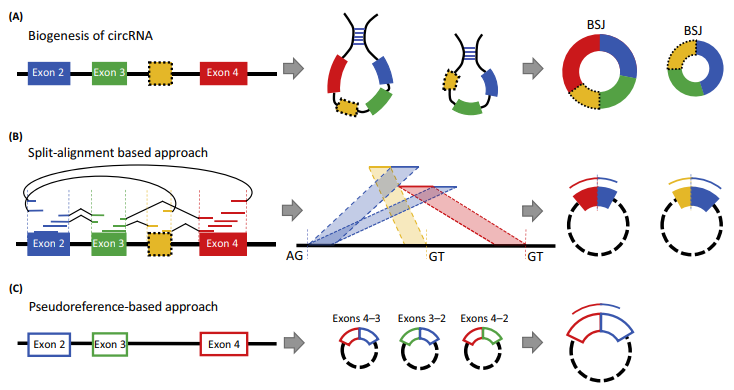
同时作者提出基因注释的使用对分析结果是有益的。注释有助于提高结合识别与从头检测的灵敏度。作者提出不同的识别软件使用不同的比对工具,比如:CIRI和KNIFE选择了在大多数基于参考的DNA / RNA序列分析中广泛使用的read-mappers,如Bowtie和BWA。诸如circRNA_finder,CIRCexplorer和DCC等检测算法使用比对软件如TopHat,STAR 和Novoalign。一些检测算法可能需要多个比对软件。NCLscan 需要BWA,BLAT和Novoalign,而UROBORUS则需要Bowtie和TopHat。多软件比对的好处是这种综合分析可能结合不同比对软件的优点,但它们也不可避免地增加了安装和使用的依赖性。
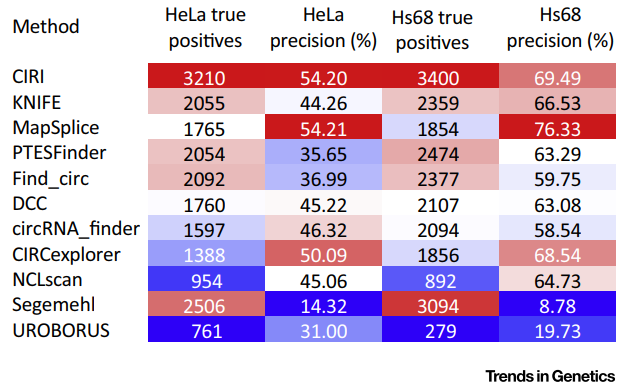
作者评估11种环形RNA(circRNA)检测方法的性能比较。
除了上面讨论的circRNA检测算法外,作者还讨论了其他计算方法,基于检测用于各种分析的circRNA,包括差异表达分析,准确定量,可选剪接检测,全长组装和可视化。
总之,自2013年以来,已发布了超过10种circRNA检测方法。这些方法中的每一种都采用不同策略的独特组合,这些策略根据基因组参考和注释使用,read比对软件的选择,BSJ read的识别而变化。具有更全面性能的检测方法将继续被开发,在这些circRNA检测方法的基础上,目前的下游工具专注于差异表达分析,准确量化,可选剪接检测,全长装配和可视化,并且将开发更多样化的下游分析方法以促进对circRNA的深入研究。
参考文献:
Gao, Y. and F. Zhao, Computational Strategies for Exploring Circular RNAs. Trends Genet,2018.
作者:计算表观遗传学 微信公众号





来第一个抢占沙发评论吧!