近日,墨西哥儿童医院的Javier T. Granados-Riveron和Guillermo Aquino-Jarquin在Biochimica et Biophysica Acta杂志撰写了综述论文,讨论了关于环状RNA表达多肽的最新发现和理论推测 (Javier T. et al. 2016)。
作者一开始着重介绍了环状RNA形成机制和作为miRNA Sponge功能以及调控转录作用的功能模型,我们在早期的一些文献跟踪报道中曾详细介绍过,在此不再赘述,感兴趣的点击 查看。接下来重点介绍了环状RNA可能直接翻译出多肽的最新发现。下面就让山人跟大家一起详细学习一下这篇综述文章吧。
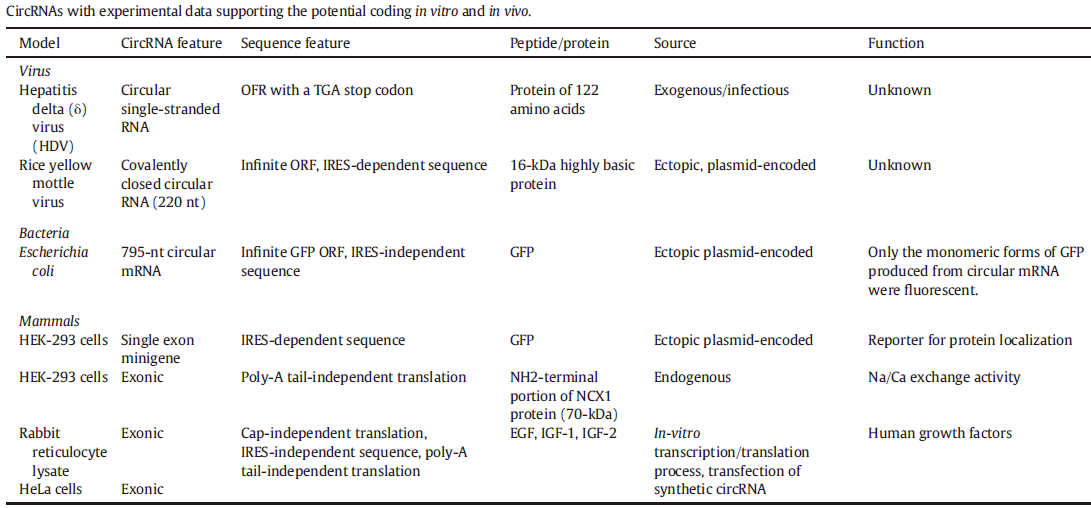
图1 目前已知的环状RNA表达多肽的证据(来自(Javier T. et al. 2016))
早在1984年,H. SCHELLEKENS等就曾在Nature上发表文章发现hepatitisδ virus(HDV)病毒中包含着单链环状RNA,并且该环状RNA可以表达一条122个氨基酸的多肽。这是人类关于环状RNA表达多肽最早的文献报道。
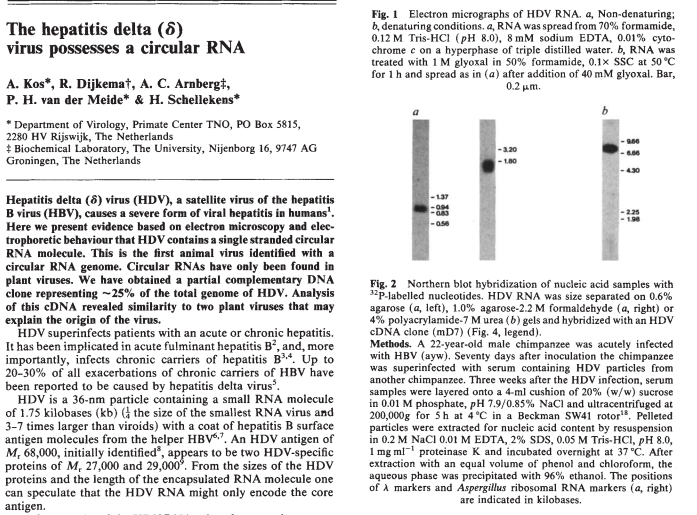
图2 HDV病毒的单链环状RNA表达多肽 (来自(Zheng et al., 2016))
2014年,Tauqeer Ahmad发表于PNAS的一篇文章证明在水稻中被一种220nt的环状RNA构成的拟病毒感染后会在这种可复制的环状RNA分子上表达出一个16kD大小的蛋白分子。

这种称为rice yellow mottle virus (sobemovirus) (RYMV) 的拟病毒可造成水稻黄斑病变。该拟病毒仅由220nt的闭合环状RNA构成。RYMV编码蛋白的机制非常奇特,我们可以初步估算一下就会发现其中玄妙之处:如果220nt全部作为密码子,最多可编码73个氨基酸,按照每个氨基酸分子量110估计,73个氨基酸最多只有8kD左右大小,实际情况是该拟病毒编码了一中16kD的蛋白!玄妙之处在于它采用了一种重叠编码的策略,就是在第一次走完环状RNA全部序列之后在起始密码子前的一个碱基和起始密码子的“AU”继续作为ORF的密码子进行第二轮编码,再次到达起始位点的时候,原先起始密码子的“A”与前面的两个碱基“UG”构成了第一终止密码子,后面还有第二和第三终止密码子存在,因此个别情况下还会表达出18kD或23kD大小的蛋白。这一文章的发现非常奇特,也只有在环状的分子形式才可能出现重叠密码子的情况。
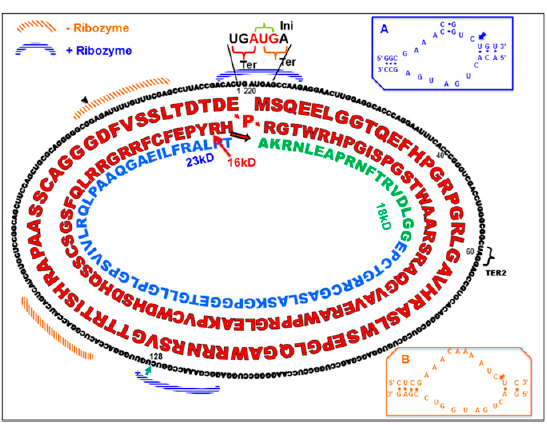
图3 RYMV环状RNA表达蛋白采用重叠密码子形式(来自(AbouHaidar et al., 2014))
在高等真核生物中除了常规的基于5’-Cap结构的蛋白翻译起始机制外,一些经常存在于病毒中的Internal Ribosomal Entry Sites (IRES)元件也可以直接启动核糖体的组装和翻译起始。有几篇文章就曾在体外和体内设计的体系中验证过有IRES元件的环状RNA可以启动蛋白翻译作用。包括Wang ZeFeng在2015年发表于RNA杂志的文章(Wang and Wang, 2015),介绍人工构建的含有IRES元件的反向拼接形成的环状RNA可表达完整的GFP蛋白。
此外,Sarnow, P.在1995年发表于Science的文章也在体外证明携带IRES元件的环状RNA能正确表达多肽(Chen and Sarnow, 1995)。Ares, M在1998年发表于RNA杂志的文章也证明在大肠杆菌中环状RNA也可以正确表达GFP蛋白(Perriman and Ares, 1998)。Abe, H.等也曾于2015年在Scientific Reports杂志发表文章证明体外可以实现不携带任何翻译起始调控元件(如IRES或Kozak序列等)的环状RNA就可以在兔网织红细胞体外翻译体系中实现ORF的翻译过程(Abe et al., 2015)。这些研究工作都初步表明环状RNA完全可以与线性DNA一样具有翻译蛋白的作用。
有趣的是,Jonathan Lytton曾于1999年在JBC杂志发表文章表明发现NCX1基因的2号外显子存在单独环化的环状RNA,该环状RNA大小1799nt,包含了NCX1基因起始密码子上游33nt的序列,由该起始密码子的ORF经过环化后正好在环化位点后面8-10位位置形成了终止密码子。因此作者推测该环状RNA能独立表达600个氨基酸构成的分子量大小约70kD的蛋白。过表达该蛋白证明可以有活性,但作者未能在体内体系中鉴定到70kD的蛋白存在。(Li and Lytton, 1999)。

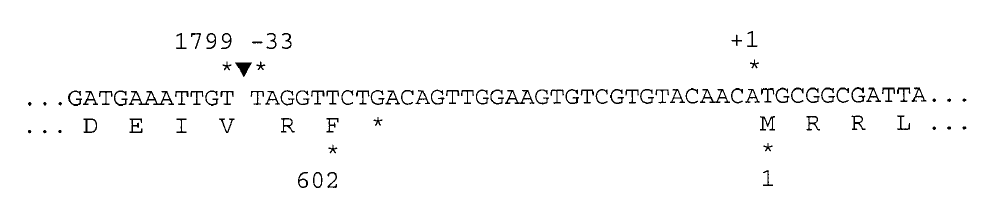
图 4 NCX1二号外显子环化后形成可表达蛋白的独立ORF (来自(Li and Lytton, 1999))
非编码RNA往往不具备典型的蛋白翻译调控序列,但一系列的研究表明一些RNA分子虽然被定义为非编码RNA,但还是有足够的证据证明可以编码蛋白和多肽的。UCSF的Jonathan S. Weissman教授先后开发了一系列鉴定具备编码功能的RNA序列的方法体系,他们曾在2009年在Science杂志发表了一种基于分离80S核糖体保护的RNA片段深度测序的方法体系,用于鉴定能够结合核糖体的RNA序列(Ingolia et al., 2009)。借助该技术,他们于2011年在Cell发表文章,发现了一些多顺反子形式的核糖体相关编码小肽的RNA(sprcRNAs)(Ingolia et al., 2011)。2013年,Eric S. Lander发表于Cell的文章提出了ribosome release score (RRS)的鉴定是否真实的进行核糖体翻译作用的评价指标(Guttman et al., 2013)。还有一些其他的作者也曾尝试找到环状RNA直接表达多肽的证据,但暂时还没有太有说服力的研究结论。

作者还介绍了几个有用的分析环状RNA翻译功能和ORF的信息学工具:ORF Finder:可用于分析核酸序列中可能存在的ORF。IRESite可用于分析目标序列中可能存在的IRES元件。CPAT可用于评估RNA编码多肽的可能性。Pfam 29.0可用于评估预测的多肽序列的基本特征。PhyloCSF可用于相关的进化分析。CircInteractome可用于分析目标环状RNA相互作用的miRNA或蛋白及其作用位点。
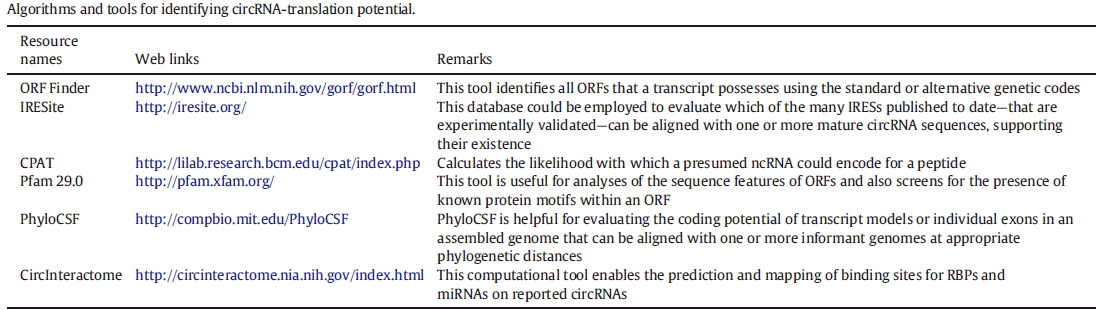
图5 有用的预测分析环状RNA表达多肽的工具 (来自(Javier T. et al. 2016))
总之,目前在哺乳动物中还没有直接的证据表明环状RNA可以翻译蛋白,但一系列的线索又非常支持这一假设。就在前几天召开的环状RNA研究论坛上,暨南大学的张弓教授也在他们翻译组学的分析平台中找出了一些环状RNA表达多肽的线索,相关结果暂未发表。这些线索和相关研究背景非常支持环状RNA会表达多肽的假设,各位同行可以针对自己感兴趣的环状RNA进行这方面的分析预测,或许会有意外的惊喜。
Javier T. Granados-Riveron, Guillermo Aquino-Jarquin. (2016). The complexity of the translation ability of circRNAs. Biochimica et Biophysica Acta. Published Online.
Abe, N., Matsumoto, K., Nishihara, M., Nakano, Y., Shibata, A., Maruyama, H., Shuto, S., Matsuda, A., Yoshida, M., Ito, Y., et al. (2015). Rolling Circle Translation of Circular RNA in Living Human Cells. Sci Rep 5, 16435.
AbouHaidar, M.G., Venkataraman, S., Golshani, A., Liu, B., and Ahmad, T. (2014). Novel coding, translation, and gene expression of a replicating covalently closed circular RNA of 220 nt. Proc Natl Acad Sci U S A.
Chen, C.Y., and Sarnow, P. (1995). Initiation of protein synthesis by the eukaryotic translational apparatus on circular RNAs. Science 268, 415-417.
Guttman, M., Russell, P., Ingolia, N.T., Weissman, J.S., and Lander, E.S. (2013). Ribosome Profiling Provides Evidence that Large Noncoding RNAs Do Not Encode Proteins. Cell 154, 240-251.
Ingolia, N.T., Ghaemmaghami, S., Newman, J.R.S., and Weissman, J.S. (2009). Genome-Wide Analysis in Vivo of Translation with Nucleotide Resolution Using Ribosome Profiling. Science 324, 218-223.
Ingolia, N.T., Lareau, L.F., and Weissman, J.S. (2011). Ribosome profiling of mouse embryonic stem cells reveals the complexity and dynamics of mammalian proteomes. Cell 147, 789-802.
Li, X.F., and Lytton, J. (1999). A circularized sodium-calcium exchanger exon 2 transcript. J Biol Chem 274, 8153-8160.
Perriman, R., and Ares, M. (1998). Circular mRNA can direct translation of extremely long repeating-sequence proteins in vivo. Rna 4, 1047-1054.
Wang, Y., and Wang, Z. (2015). Efficient backsplicing produces translatable circular mRNAs. RNA 21, 172-179.
Zheng, Q., Bao, C., Guo, W., Li, S., Chen, J., Chen, B., Luo, Y., Lyu, D., Li, Y., Shi, G., et al. (2016). Circular RNA profiling reveals an abundant circHIPK3 that regulates cell growth by sponging multiple miRNAs. Nat Commun 7, 11215.





来第一个抢占沙发评论吧!