转自: 生信菜鸟团(微信公众号)
上一期我们说到,使用不同的生物信息工具分析同一组数据可能获得不同的结果,自然而然出现了一些评估程序的文章。简单重复一下主要结论,在circRNA数据分析中,测序文库需要RNase 处理;面对质量不高的数据,应对数据过滤后再进行分析;有参优于 De Novo分析;结合各个识别程序的优势,取两个以上程序的鉴定结果交集,会进一步降低预测的假阳性。今天的推文带来武器库中的第二把利器:KNIFE。

已有多篇报道评估,KNIFE这么稳定输出的程序我们来看一下究竟。
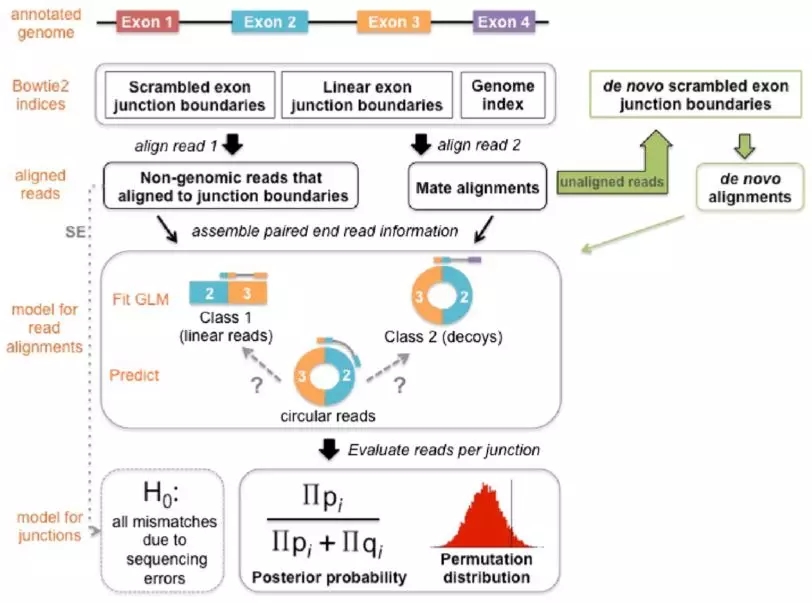
原理简述
使用统计学模型提高对RNA-seq数据circRNA分析的敏感性和特异性。对于PE数据,使用 Bowtie2 分别独立map到genome, ribosomal RNA, linear exon–exon junctions, and scrambled exon–exon junctions。被标记为 “junctional” 的读段,read1 (R1)必须覆盖设定的核苷酸数量,且并未比对到基因组和核糖体组。假阳性有以下两种情况:
1、those that map to canonical linear isoforms (“real” alignments)
2、those which are likely artifacts because the relative alignment orientations of the paired-end reads are consistent with neither a linear nor a circular RNA (“decoy”
alignments)
即一是比对到了线性转录本上,二是指比对上的序列方向是一致的。
为了应对 decoy reads,作者拟合了一个logistic generalized linear model (GLM),参数包含 alignment score (a composite of sequencing quality and mismatchrate), mapping quality, and offset position,并计算真阳性概率,最终获得的 cirular junctions 超过90%与decoy reads较低关联。
流程介绍
根据介绍,程序被包装到一个shell脚本中,这个轮子还是很好用的。
1、首先根据要求构建分析环境
需要注意的切换 python2 的使用环境,bowtie 主要用于 de novo 分析流程中。

2、从github上获取基本信息,由于开发板还存有一些bug,因此选择v1.4稳定版内容
$ wget https://github.com/lindaszabo/KNIFE/archive/v1.4.tar.gz
$ tar zxvf v1.4.tar.gz
# 添加执行权限,否则运行过程中会由于部分软件无执行权限报错。
$ cd KNIFE-v1.4/circularRNApipeline_Standalone/analysis
$ sudo chmod a+x *
3、根据作者提供的数据进行测试,作者将构建好的索引打包至 MEGA 网盘,链接如下:https://mega.nz/#F!RtsCHCQb!fyxYNWjoCef5Ie361vUxiA。亲测可用,下载平均速度在5m/s。随后下载测试数据,测试数据为人类样本,因此索引下载hg19。
# 将下载好的索引移至一下位置
$ cd circularRNApipeline_Standalone/denovo_scripts/index
$ tar zxvf hg19_BT1index.tar.gz
# 并将hg19的gtf注释文件移入以下的路径中
$ cd circularRNApipeline_Standalone/index
$ tar zxvf hg19_BT2index.tar.gz
# 下载测试数据
$ cd circularRNApipeline_Standalone/testData
$ wget -c https://github.com/lindaszabo/KNIFE/releases/download/v1.1/SRR1027187_*.fq.gz
4、测试
$ cd circularRNApipeline_Standalone
$ sh completeRun.sh ./testData/ complete ./new testData 8 phred64 circReads 40 2>&1 | tee out.log
结果文件应该包含
- SRR1027187_1_linearJuncProbs.txt and SRR1027187_1_circJuncProbs.txt
- SampleAlignStats.txt and SampleCircStats.txt
同时,作者提供自建索引的方法,可以详见 INSTALL。就酱。



来第一个抢占沙发评论吧!