微信:
撰文丨黄蔚
责编丨迦溆
环状RNA(circular RNA,circRNA)是一类呈环形闭合的单链非编码RNA分子,不具有5‘末端帽子和3’末端多聚腺苷酸化(poly A)尾巴结构。
早在1979年,Hsu MT等人通过电子显微镜的观察,首次在真核细胞中发现了环状RNA【1】。但当时的观点认为此类RNA属于转录的“噪声”,因而没有得到重视。
随着生物信息学的发展,大量长链非编码RNA(长度大于200nt)被鉴定,引起了广泛的关注。然而,主流的转录组分析大多基于poly A富集的RNA,因此,环状RNA虽然与长链非编码RNA一样广泛存在,但却未被发掘。
直到2013年,Sebastian Memczak等人利用去除rRNA的转录组数据分析【2】,鉴定了大量环状RNA,并发现环状RNA可通过吸附miRNA发挥功能的作用方式。之后的5年间,环状RNA几乎成为非编码RNA研究领域最热门的选手之一,环状RNA被广泛地鉴定在多种体系中存在,与癌症发生发展的关系也越来越明确。
然而,关于转录组中究竟有多少环状RNA,这个最基本的问题,一直没有得到很好的回答。基于芯片的环状RNA表达谱分析,只能了解已鉴定的环状RNA。转录组分析可以鉴定新的环状RNA,但目前主要是去除rRNA (RiboZero) 以及RNase R处理(RNase R属于核酸外切酶,可消化线性RNA)后的转录组分析方法,前者需要大于5ug的RNA量,后者由于缺乏线性RNA,无法比较环状RNA和产生它的线性RNA的丰度。
同时,关于环状RNA在癌症中的作用方式,大量研究发现环状RNA通过竞争性内源RNA(ceRNA)的作用方式,即吸附miRNA,使miRNA无法靶向原目标基因的3‘UTR,发挥功能。由于环状RNA对miRNA的吸附仅需要7-9nt的碱基互补配对即可,ceRNA理论似乎成为了处理环状RNA作用机制的“捷径”。环状RNA结合miRNA后,是否真的解除了miRNA对原靶基因的抑制呢?目前这一理论已受到挑战。
2月7日,Cell同时在线了两篇环状RNA的研究,试图为我们解开以上2个疑问,或者说,他们至少让我们离“真相”更近。
1、关于转录组中环状RNA的数量。来自美国密歇根大学的Arul M. Chinnaiyan教授等发表研究The Landscape of Circular RNA in Cancer,首次利用外显子捕获技术在超过2000例癌症临床样品中,得到了最全的环状RNA表达谱,并鉴定了一类由两个基因的外显子组成的新型环状RNA——Read-through circRNA (图1)。

图、1
由于目前的研究发现大部分环状RNA都来源于基因的外显子【2】,研究者利用探针捕获已知的外显子序列,再进行深度测序。通过这样的方式,研究者在超过10种癌症的2000例临床样品中,鉴定到了超过3000个环状RNA。与传统的RiboZero处理得到的环状RNA相比,外显子捕获测序(Capture)得到的环状RNA更多,同时,也包含了大部分RiboZero中的环状RNA,说明Capture方法可以更有效的鉴定环状RNA(图2)。同时,由于该方法保留了线性RNA,研究者还发现环状RNA的表达量与母基因(parent gene)产生的mRNA数量没有明显的关系,因此不能简单地将环状RNA表达量的变化归结为母基因表达的mRNA的变化。
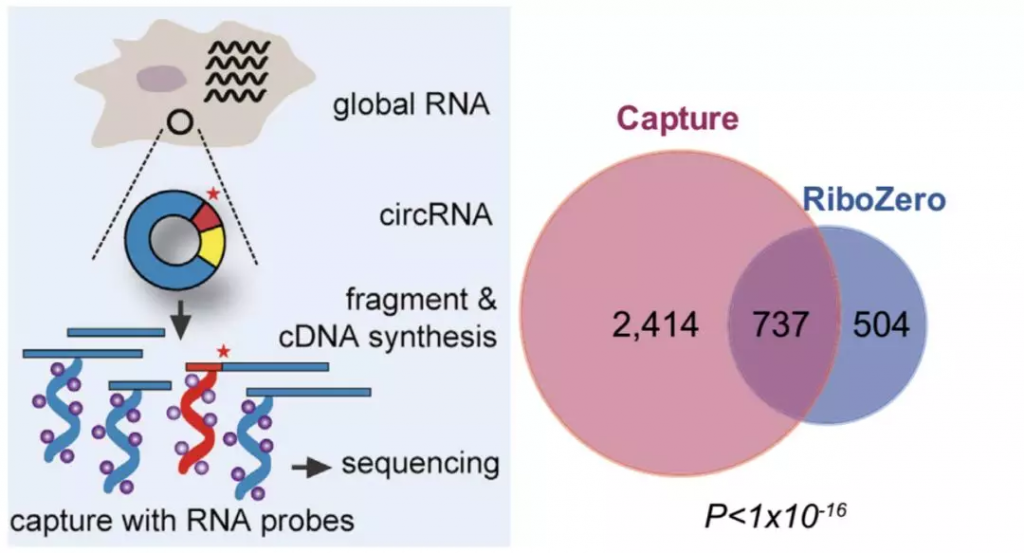
图、2
值得注意的是,研究者将以上环状RNA的分析结果整合到了MiOncoCirc数据库中,这是一个面向所有科研工作者的目前最全面的癌症中环状RNA鉴定和表达模式分析网站,不仅包含环状RNA的基因座位信息,还有表达量、基因组结构以及临床样品的数据(图3)。
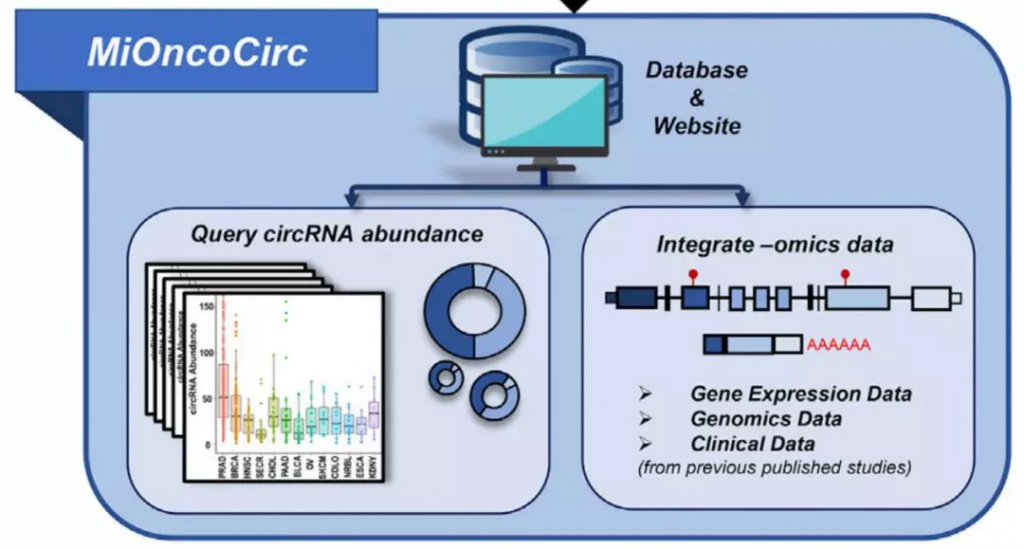
图、3
有趣的是,研究者通过成对的癌组织与正常组织分析发现,环状RNA在癌症中的表达量普遍低于正常组织,并鉴定了一批与预后相关的环状RNA。环状RNA具有较高的稳定性,这提示它们有作为生物标志物的潜力。非入侵式检测是一种理想的临床检测手段,研究者通过在尿液中提取的仅50ng的RNA,采用外显子捕获的方式鉴定了1092个环状RNA。这说明利用外显子捕获技术对环状RNA进行鉴定具有重要的临床意义。
此外,在大量的数据分析中,研究者鉴定了一类由两个基因的外显子组成的新型环状RNA,命名为:Read-through circRNA(rt(-circRNA)。不同于Guarnerio等人2016年也在Cell发表的f-circRNA——来源于融合基因的融合位点两侧外显子的环状RNA,rt-circRNA由位于同一条DNA链的两个临近基因的外显子组成(事实上,该研究在2000例样品的分析中都没有发现f-circRNA的存在)。并且rt-circRNA占所有环状RNA的2.5%,数量并不少。它们的形成与RNA聚合酶II在基因座位上的通读(Read-through)相关(图4)。
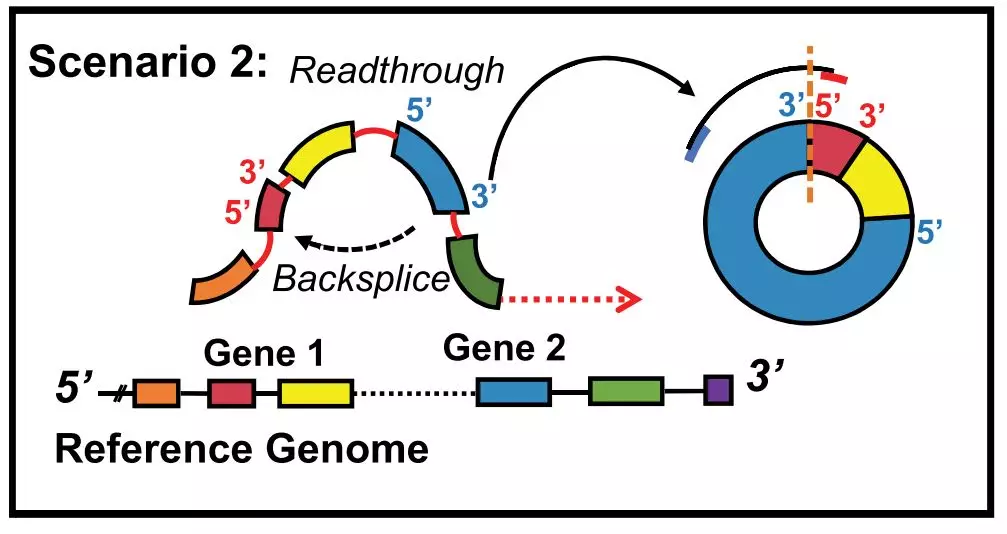
图、4
总的来说,该研究建立了目前最全面的癌症中环状RNA表达谱分析网站,为环状RNA的鉴定及它们在癌症中的功能研究奠定了基础。但同时也应注意,环状RNA也可由内含子组成,而该研究采用的外显子捕获方法,显然错过了这一部分环状RNA,因此,基因组中的环状RNA究竟有多少,还需要更深入的研究。
2、环状RNA结合miRNA的生理意义。来自多伦多大学的Housheng Hansen He教授等发表研究Widespread and Functional RNA Circularizationin Localized Prostate Cancer,首次在局限性前列腺癌中系统的鉴定了环状RNA的表达和功能(图5)。


图、5
局限性前列腺癌是一类常见的男性癌症,可通过明确的局部治疗得到完全缓解,而一旦癌细胞向外转移,则发展为难以治愈的高侵袭性癌症。因此,了解局限性前列腺癌发展的生物学过程,是非常必要的。研究者拟从环状RNA的角度分析前列腺癌的发展过程。在 144例前列腺癌临床样品的去除rRNA转录组数据中,研究者共鉴定了76311个环状RNA,并发现它们的表达模式与癌细胞的侵袭性相关。更重要的是,利用shRNA文库的筛选,研究者在1336个高表达的环状RNA中,鉴定了171个环状RNA与细胞生长相关,而敲低这些环状RNA对应的mRNA则不影响细胞增殖。该结果说明环状RNA在前列腺癌的发展中发挥重要作用,并且它们的作用机制独立于母基因转录的mRNA。
那么,环状RNA是如何行使功能的呢?考虑到环状RNA可通过结合miRNA发挥功能【3】,研究者以能促进前列腺癌细胞增殖的circCSNK1G3为例,分析了可能与它结合的miRNA。通过miRNA表达谱及AGO2-clip seq数据分析,发现circCSNK1G3可能通过结合miR-181b/d发挥功能(图6)。
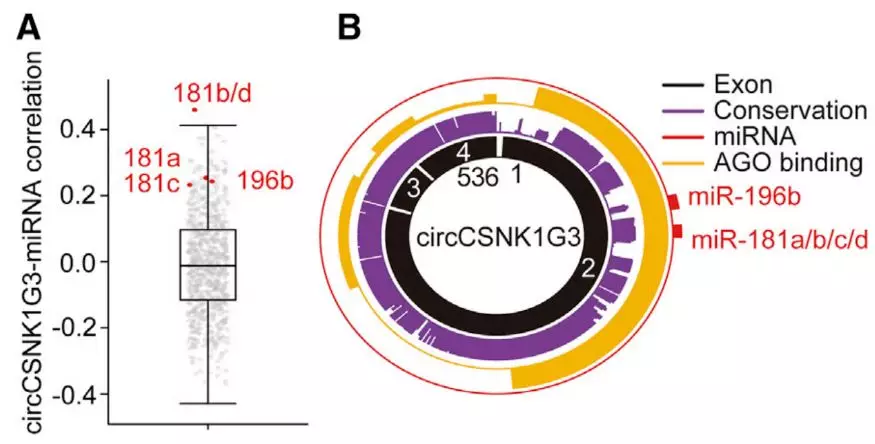
图、6
有趣的是,敲低circCSNK1G3对miR-181b/d靶基因(CBX7,CDK1和CDC25A)的影响与过表达miR-181b/d相反,并且回复实验显示在敲低circCSNK1G3后过表达miR-181b/d反而能促进细胞的增殖,这说明circCSNK1G3与miR-181b/d在前列腺癌中都发挥癌基因的作用,circCSNK1G3结合miR-181b/d后并没有抑制miR-181b/d的活性,反而circCSNK1G3的减少导致miR-181b/d无法抑制靶基因的表达。这与常见的环状RNA的ceRNA作用方式不符。
其实,早在2017年,Piwecka等人在Science上发表过类似现象的研究【4】,他们认为环状RNA吸附miRNA后,实际上是稳定了miRNA,使得miRNA可以更好的抑制靶基因的表达,而环状RNA的缺失会造成miRNA的降解,因此此时靶基因的表达量反而会上升。而circCSNK1G3的研究正好与它不谋而合。
总的来说,上述研究首次系统的鉴定了前列腺癌中环状RNA的表达,并发现了促进癌症发展的环状RNA。但是,显然地,他们的研究忽略了我们上文提到的rt-circRNA,因此,想要全面的了解前列腺癌中环状RNA的表达谱,还需进一步分析。同时,我们还应注意到,环状RNA结合miRNA后,对miRNA命运的决定具有多样性,既可能抑制miRNA的活性,也可能促进miRNA的稳定,而这两种完全不同的作用方式是如何被调控的,目前还不得而知。该问题的解决将有利于我们更全面的了解环状RNA的作用机制。
参考文献
1. Hsu MT, Coca-Prados M. Electron microscopic evidence for the circular form of RNA in the cytoplasm of eukaryotic cells. Nature. 1979 Jul 26;280(5720):339-40.
2. Memczak, S., Jens, M., Elefsinioti, A., Torti, F., Krueger, J., Rybak, A., Maier,L., Mackowiak, S.D., Gregersen, L.H., Munschauer, M., et al. (2013). CircularRNAs are a large class of animal RNAs with regulatory potency. Nature 495,333–338.
3. Hansen, T.B., Jensen, T.I., Clausen, B.H., Bramsen, J.B., Finsen, B., Damgaard, C.K., and Kjems, J. (2013). Natural RNA circles function as efficient microRNA sponges. Nature 495, 384–388.
4. Piwecka, M., Glazar, P., Hernandez-Miranda, L.R., Memczak, S., Wolf, S.A.,Rybak-Wolf, A., Filipchyk, A., Klironomos, F., Jara, C.A.C., Fenske, P., et al.(2017). Loss of a mammalian circular RNA locus causes miRNA deregulationand affects brain function. Science 357, eaam8526
制稿人:子阳





来第一个抢占沙发评论吧!